更快的R:一个实例
最近玩 R,出现了很多有意思的画面,今天来复盘一到错题,温故知新。初始问题是这样的:帮忙看看用户的购买间隔,为沟通机制提供数据参考。🤔️ 开动,38万行数据,第一锤子下去,敲出来的代码如下:
library(tidyverse)
library(lubridate)
data %>%
filter(created_at >= '2020-07-01',
created_at < '2021-01-01',
is.na(refund_state)) %>%
select(member_no, union_no, created_at) %>%
mutate(created_at = date(created_at)) %>%
group_by(member_no, union_no) %>%
slice_max(created_at, n = 1) %>%
ungroup() %>%
select(-union_no) %>%
distinct() %>%
group_by(member_no) %>%
arrange(created_at) %>%
mutate(
n_days = n(),
tier = case_when(
n_days == 1 ~ "1d",
n_days == 2 ~ "2d",
n_days == 3 ~ "3d",
n_days > 3 ~ "3d+"),
previous = lag(created_at),
dd = interval(previous, created_at) %/% days(1))
使用 tidyverse 语法的好处之一就是易读,这里的逻辑是这样的:
filter筛选必要的行select筛选必要的列mutate更改日期时间为仅日期(间隔日,不需要理会时间点)group_by按用户和单号进行分组filter同一单选最大日期(发现同一单有时间不一致的情况)ungroup取消分组select丢掉单号列distinct针对剩下的用户id和下单日期,去重group_by再次按用户id分组arrange按日期在分组内排序mutate计算活跃(购买)天数并按天数分层lag找出当前日期的上一个日期interval计算两个日期之间的间隔
转换思路
跑跑看,猜怎么着?看起来好像没啥问题,跑起来慢的要死,两分钟没有结束迹象,这种情况,显然是哪里犯傻了,得先揪出明显的性能瓶颈再说。这时候 tidyverse 语法的另一个好处又体现出来了,它的代码组织形式,对于一行行或一块块的问题排查,非常友好。我们可以先采用二分法,以 ungroup 为界,先看上半部分。到底慢在哪里,我们可以借助 profvis 这个工具来分析,把需要检测的代码塞到 profvis({...}) 内部即可。上半部分的结果如下图
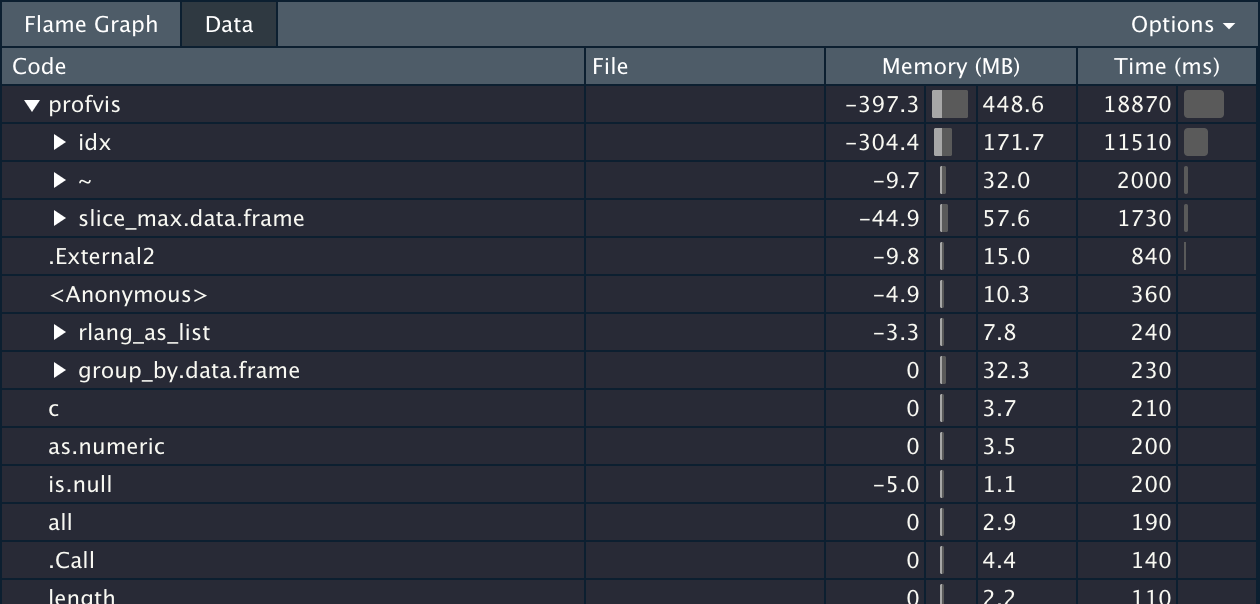
slice_ 和时区
直接使用 slice_max 筛选最大值是不合适的,消耗了 11 秒多,先 arrange 对这两个关键列进行排序,再 slice_head/tail 会快不少,但它显然不是大头,这十来秒不会是漫长无应答的主要原因。
另外,我们的目的,只是为了获得去重的用户id和下单日期,引入 union_no 单号似乎毫无必要,但是这一顿操作只是为了修正一单跨两个日期的情况;但话又说回来,虽然发现存在一单有多个时戳的情况,到底跨没跨日期,其实可以另行探查;而探查的结果是,没有 😂️,所以上半部分的最终的改进结果如下:
data %>%
filter(created_at >= '2020-07-01',
created_at < '2021-01-01',
is.na(refund_state)) %>%
select(member_no, created_at) %>%
mutate(created_at = date(created_at)) %>%
distinct()
# ...
上半部分,就只是一百毫秒左右的事情了。但是问题又来了,发现两部电脑的运行结果不一致,还好略有经验,猜测是时区处理的问题,编辑 ~/.Renviron 文件,加入 TZ 参数即可,我的配置如下,其他选项可参考:
TZ="UTC"
RETICULATE_PYTHON="/usr/local/bin/python3"
R_MAX_VSIZE=16Gb
http_proxy=http://127.0.0.1:1091/
https_proxy=http://127.0.0.1:1091/
再来看后半部分,它只可能慢在 mutate 内部,推测着,先把 interval 这行注释掉,运行结果如下图:
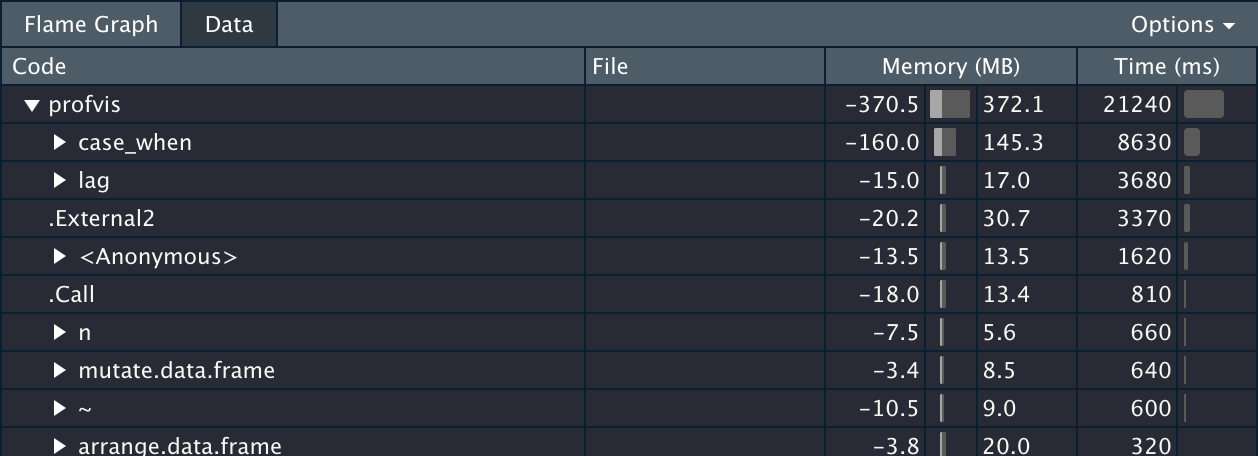
case_when 和 lag 都很慢,加起来十几秒,不过,这十几秒也让最终 boss 浮出了水面:interval 。这里其实有点奇怪,单拿出来跑, interval(df$a, df$b) %/% days(1) 是很快的,应该是嵌套到 mutate 内部之后,失去了 vectorized 特性,变成了 row-wise 的循环。改用 base R 提供的 lagged differences 算法之后,就直接光速了:
data %>%
filter(created_at >= '2020-07-01',
created_at < '2021-01-01',
is.na(refund_state)) %>%
select(member_no, created_at) %>%
mutate(created_at = date(created_at)) %>%
distinct() %>%
group_by(member_no) %>%
arrange(created_at) %>%
mutate(
n_days = n(),
tier = case_when(
n_days == 1 ~ "1d",
n_days == 2 ~ "2d",
n_days == 3 ~ "3d",
n_days > 3 ~ "3d+"
),
dd = c(0, diff(created_at)))
改动如上,原 lag 和 interval 并作一行 c(0, diff(created_at))),至此,从遥遥无期到十几秒跑完,算是一个里程碑,最后还剩下一个 case_when,一时想不出什么办法。
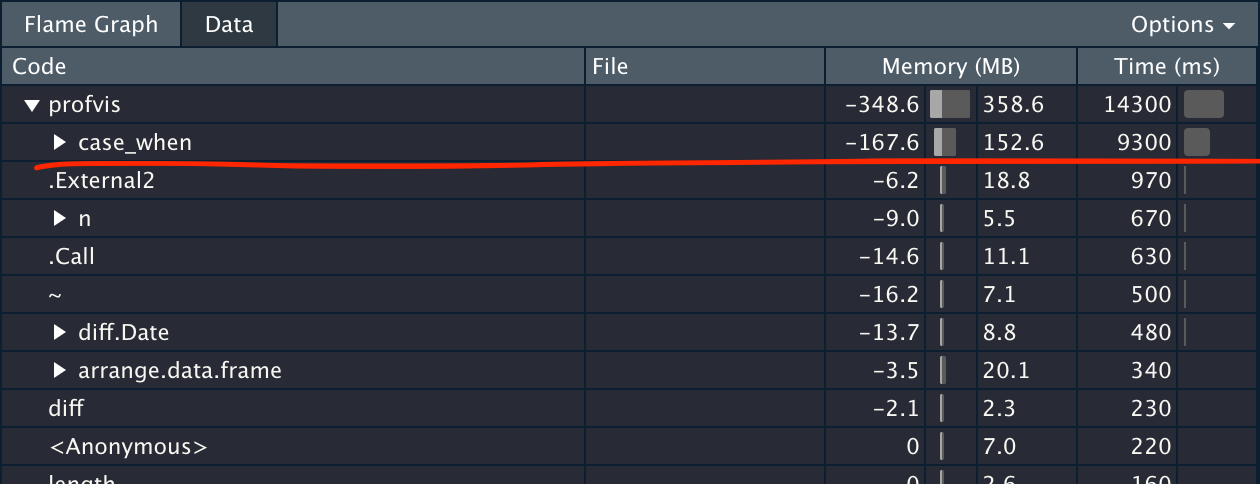
更换工具
data.table 是 R 宇宙当中以性能见长的数据处理库,它的语法抽象而简洁,几乎一句话就可以概括:DT[i, j, by]。只是一旦 chain 到一起,(至少对我来说)就有点绕毛线了,因为它的 chain 是这样的:
# data.table 原生 chain
dt[][
][
][
]
# 当然也可以使用 magrittr 的 pipe
# 但内容一多,括号里面的内容就不好分割了
dt %>%
.[] %>%
.[] %>%
.[]
tidyverse 语法虽然看似罗嗦,但是对比之下,在 base、tidy、data.table 这三大宇宙当中,一点点语法冗余,换来更好的排版和阅读便利,还是非常值得的。为了二者兼得,tidyverse 还在 dplyr 的基础上,提供了一个桥接方案 dtplyr,桥接的后端就是 data.table。它会在运行之前,先将 dplyr 语法转义为 data.table 语法,以期实现「写的舒服」「跑的还快」的双重目的。这个方案在之前的一些小测试中,我并没发现有大的性能差异,最近重建了 R 环境,较为仔细地完成了 openblas 和 libomp 的编译支持,正好一试。
使用 dtplyr 并不算麻烦,可以把 dplyr 草稿纸,注意两件事就好:一是通过 data.table 来读入数据,获得 data.table 结构;二是,操作的开头需要调用 lazy_dt 创建(或者说告知这是)一个草稿,中间 dplyr 等 tidyverse 那些逻辑正常写,结尾再调用 as_tibble 或 as.data.table 或 as.data.frame 来获得完整结果(或者说告知执行)。
dt %>% # <----
lazy_dt() %>% # <----
filter(created_at >= '2020-07-01',
created_at < '2021-01-01',
is.na(refund_state)) %>%
select(member_no, created_at) %>%
mutate(created_at = date(created_at)) %>%
distinct() %>%
group_by(member_no) %>%
arrange(created_at) %>%
mutate(
n_days = n(),
tier = case_when(
n_days == 1 ~ "1d",
n_days == 2 ~ "2d",
n_days == 3 ~ "3d",
n_days > 3 ~ "3d+"
),
dd = c(0, diff(created_at))
) %>%
as.data.table() # <----
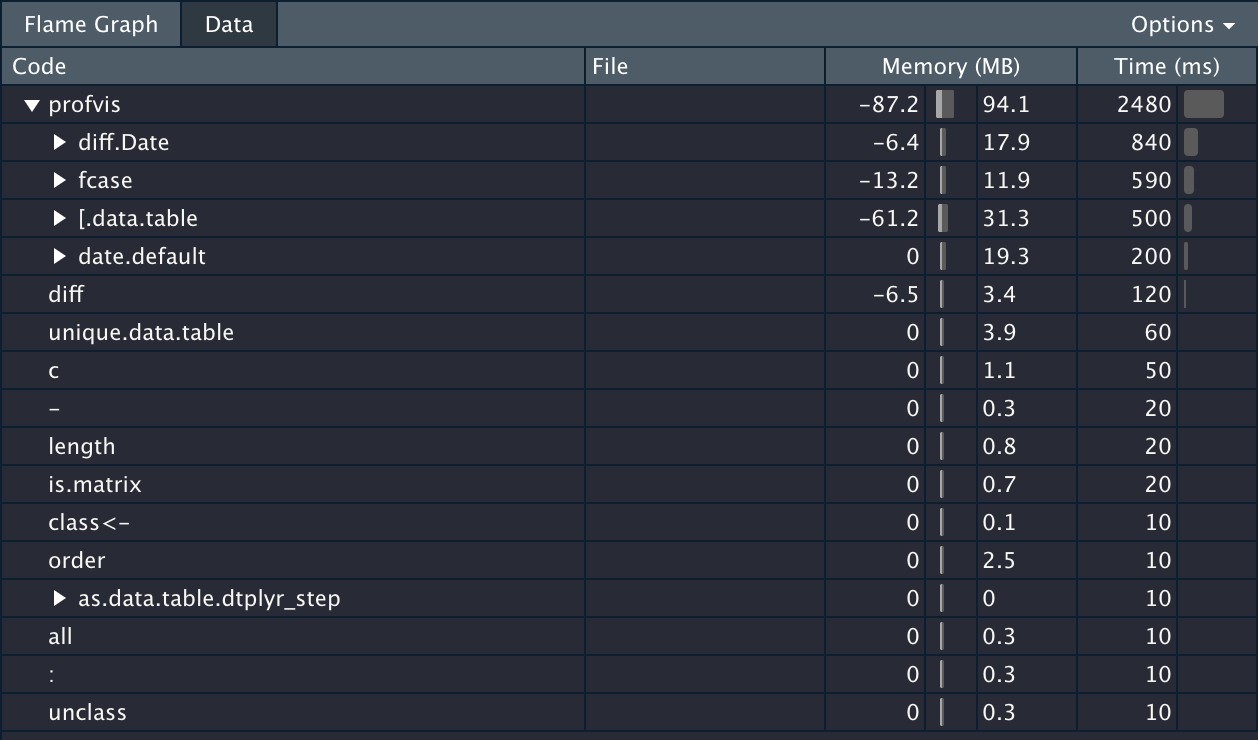
性能差异非常明显,case_when 转义成跑在 data.table 上的 fcase 之后,快了一个数量级。两个部分加一起总共耗时约两秒钟,并且注意内存那一栏,相较于 dplyr,消耗也小了很多。
# system.time({...})
user system elapsed
2.915 0.037 2.098
把末尾的 as.data.table() 替换成 show_query() 即可获得翻译结果,如下:
unique(`_DT2`[created_at >= "2020-07-01" & created_at < "2021-01-01" &
is.na(refund_state), .(member_no, created_at)][, `:=`(created_at = date(created_at))])[order(created_at)][,
`:=`(c("n_days", "tier", "dd"), {
n_days <- .N
tier <- fcase(n_days == 1, "1d", n_days == 2, "2d", n_days ==
3, "3d", n_days > 3, "3d+")
dd <- c(0, diff(created_at))
.(n_days, tier, dd)
}), by = .(member_no)]
使用 dtplyr 和 data.table 前后的火焰图,也能看得出大不同:
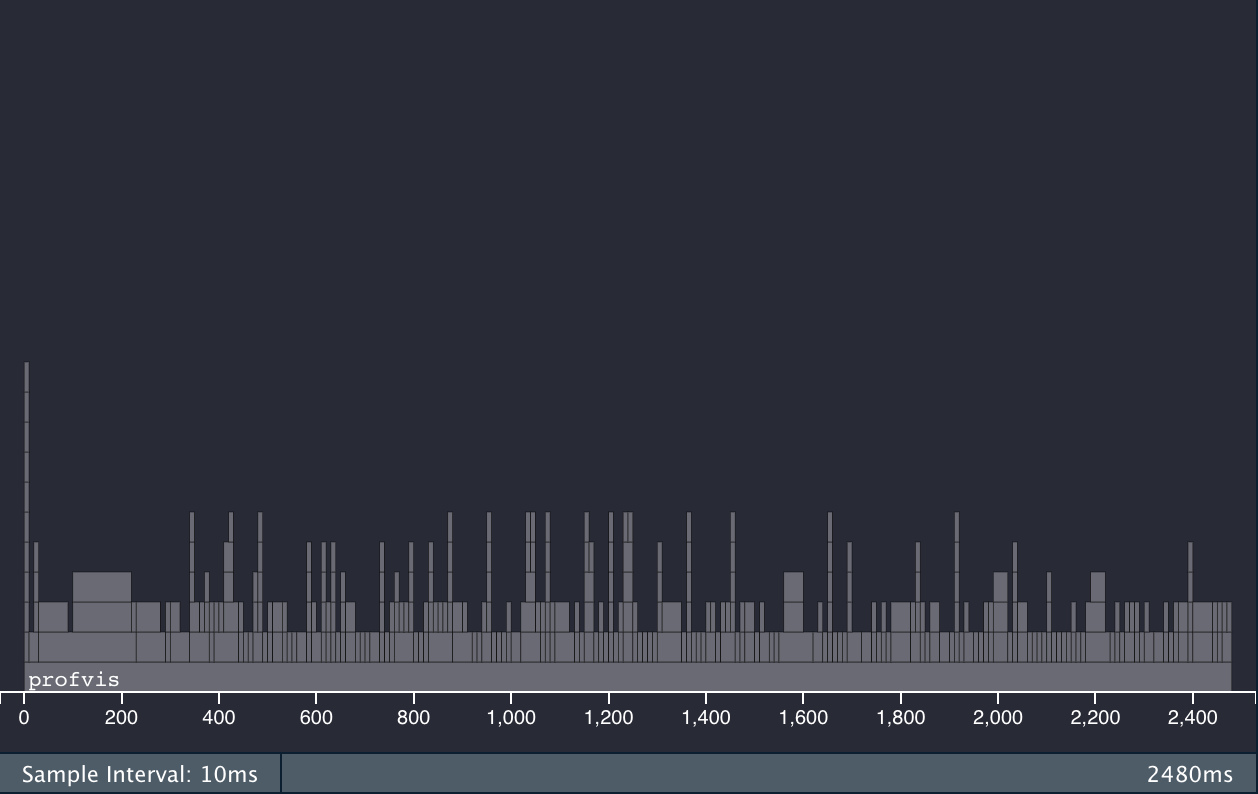 dtplyr + data.table 火焰图
dtplyr + data.table 火焰图
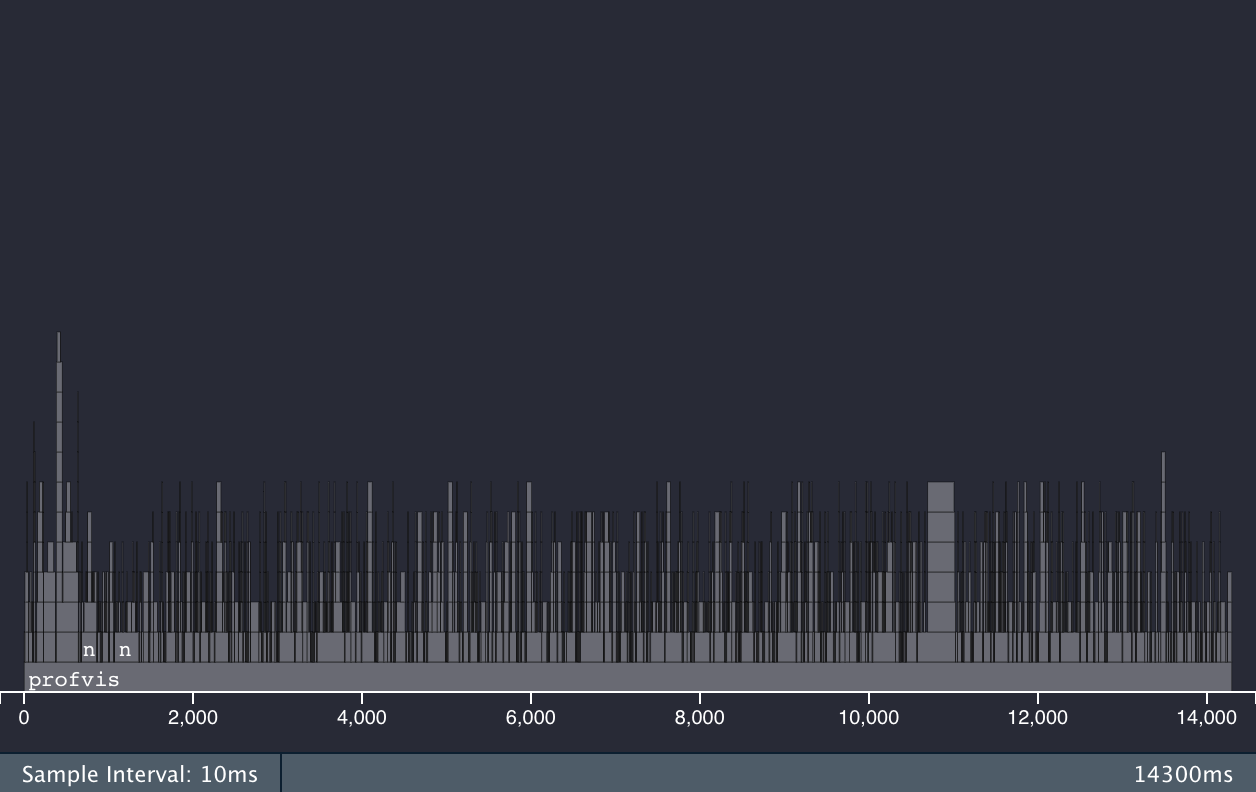 dplyr 火焰图
dplyr 火焰图
小结
Unit: seconds
expr min lq mean median uq max neval
dp 18.792854 19.282197 19.793766 19.602105 20.323187 20.96849 5
dt 2.163451 2.220804 2.235009 2.234266 2.277006 2.27952 5
# 注释掉 case_when 和 diff 两处运算之后的对比
Unit: milliseconds
expr min lq mean median uq max neval
dp 1874.7782 1932.8641 2036.7339 2041.8302 2060.804 2273.3928 5
dt 134.3752 138.8937 155.6884 141.1073 142.066 221.9998 5
对于数十万及以上规模的数据集,dtplyr + data.table 速度提升明显。
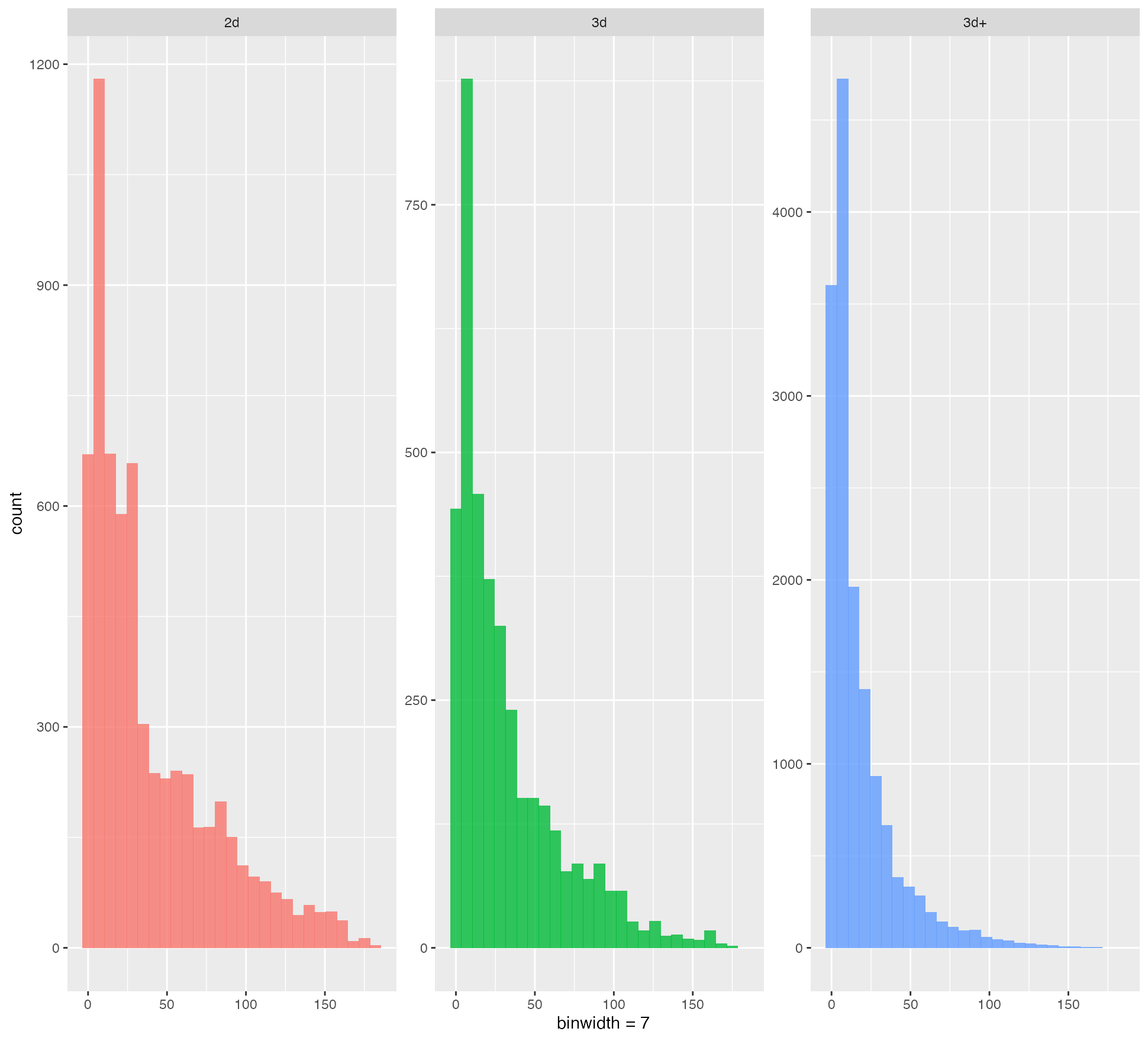 样例结果绘图
样例结果绘图